本文介绍 Bray Curtis Dissimilarity 的概念和计算方法。
Bray Curtis Dissimilarity(Bray-Curtis 相异度)是生态学中用来衡量不同样地物种组成差异的参数。
其定义和计算公式为:
\[BC_{ij}=1-2C_{ij}/(S_{i}+S_{j})\]
其中:
- \(i\) 和 \(j\) 是两个样地;
- \(S_i\) 是样地 \(i\) 中物种的总数;
- \(S_j\) 是样地 \(j\) 中物种的总数;
- \(C_{ij}\) 在两块样地中每个物种的较少计数的总和。
简单的例子
有两个水族箱:
- 一号:6条金鱼,7条孔雀鱼和4只螃蟹;
- 二号:10条金鱼和6只螃蟹。
为了计算 Bray-Curtis,首先计算\(C_{ij}\)。金鱼两个地方都有,较少的是6;孔雀鱼只在一号中有,所以不能添加;螃蟹两个地方都有,较少的是4。因此,\(C_{ij}=6+4=10\)。
\(S_i=6+7+4=17\),而\(S_j=10+6=16\)。
因此,
\[BC_{ij}=1-(2*10)/(17+16)=0.39\]
Bray-Curtis Dissimilarity 的性质
其取值介于0-1之间。如果是0,则两个样地共享所有相同的物种;如果是1,则它们不共享任何物种。
还有另外一个 Bray-Curtis index,其取值是 1 - Bray-Curtis dissimilarity。表示两个样地之间的相似程度。
适用情况
要计算 Bray-Curtis Dissimilarity,必须假设两个样地的面积或体积大小相同(与物种计数相关),否则需要在计算前调整计数。
R语言实现
vegan提供了vegdist来计算这个数值。
## 用 vegdist 重复上面的例子
df <- data.frame(goldfish=c(6,10),guppies=c(7,0),crab=c(4,6))
require("vegan")## 载入需要的程辑包:vegan## 载入需要的程辑包:permute## 载入需要的程辑包:lattice## This is vegan 2.5-6vegdist(df,method = "bray") # 这个值跟前面人脑计算的一致## 1
## 2 0.3939394## 计算群落数据
data("varespec")
## 计算不同样地两两之间的差异度
bc <- vegdist(varespec)
## 使用pheatmap做一个热图
pheatmap::pheatmap(bc,breaks = seq(0,1,0.01))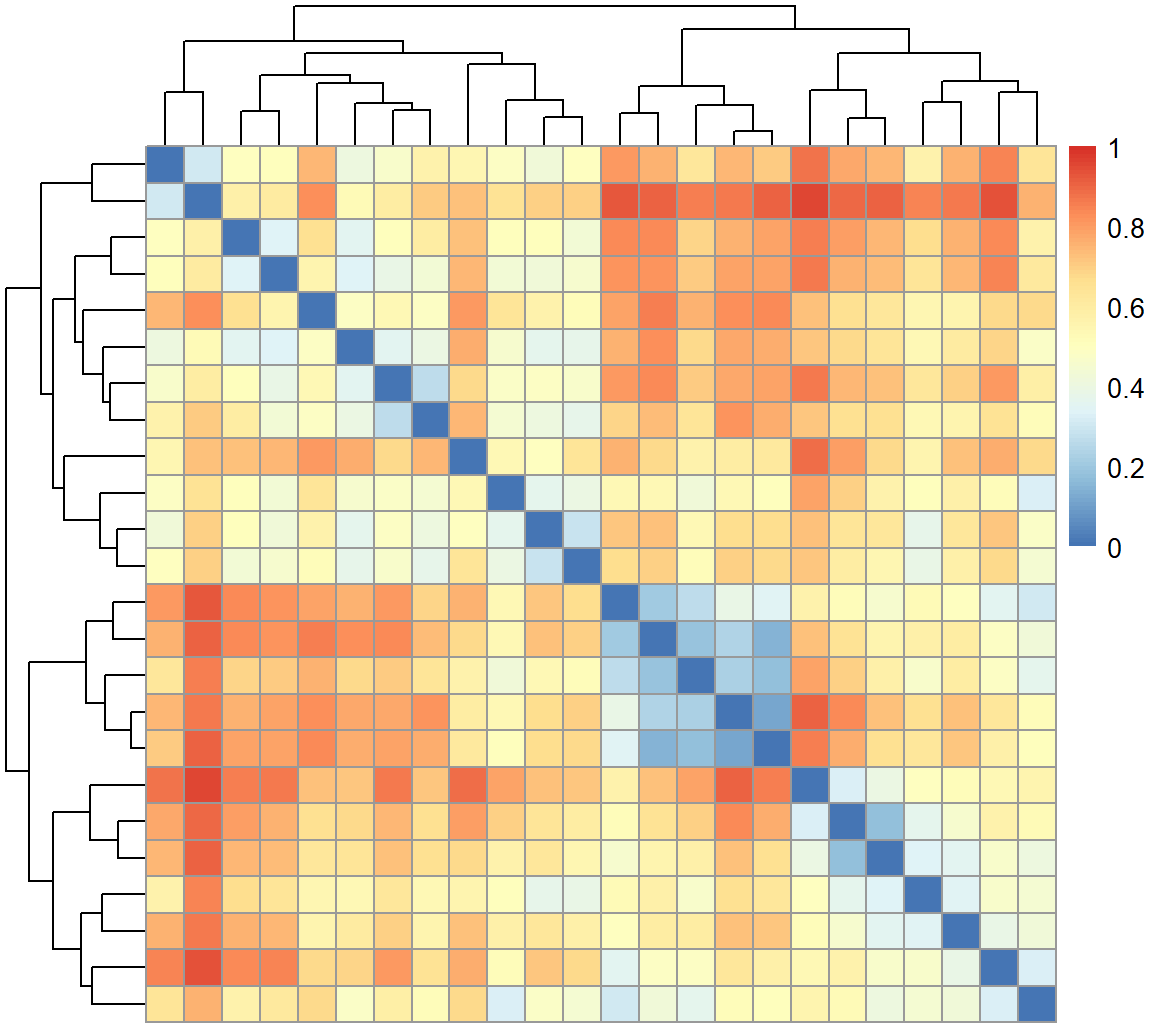
延伸阅读
vegdist的可用的方法(method = "bray")还包括 “manhattan”, “euclidean”, “canberra”, “clark”, “bray”, “kulczynski”, “jaccard”, “gower”, “altGower”, “morisita”, “horn”, “mountford”, “raup”, “binomial”, “chao”, “cao” or “mahalanobis”。
designdist可用来设计自己的差异度算法。